# ANIclustermap



[](https://pypi.python.org/pypi/aniclustermap)
[](https://anaconda.org/bioconda/aniclustermap)
## Overview
ANIclustermap is easy-to-use tool for drawing ANI(Average Nucleotide Identity) clustermap between all-vs-all microbial genomes.
ANI between all-vs-all genomes are calculated by [fastANI](https://github.com/ParBLiSS/FastANI)
(or [skani](https://github.com/bluenote-1577/skani)) and clustermap is drawn using seaborn.
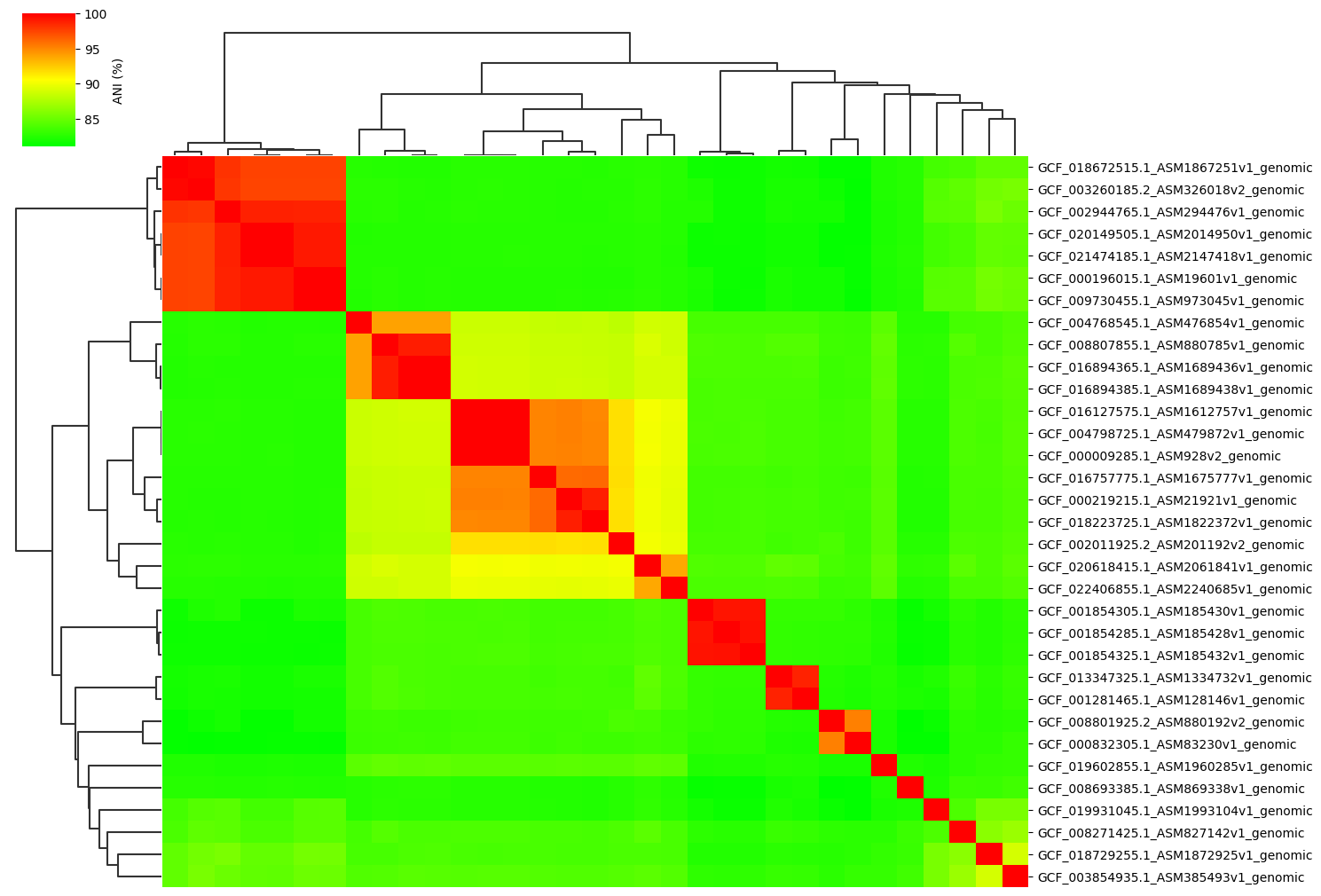
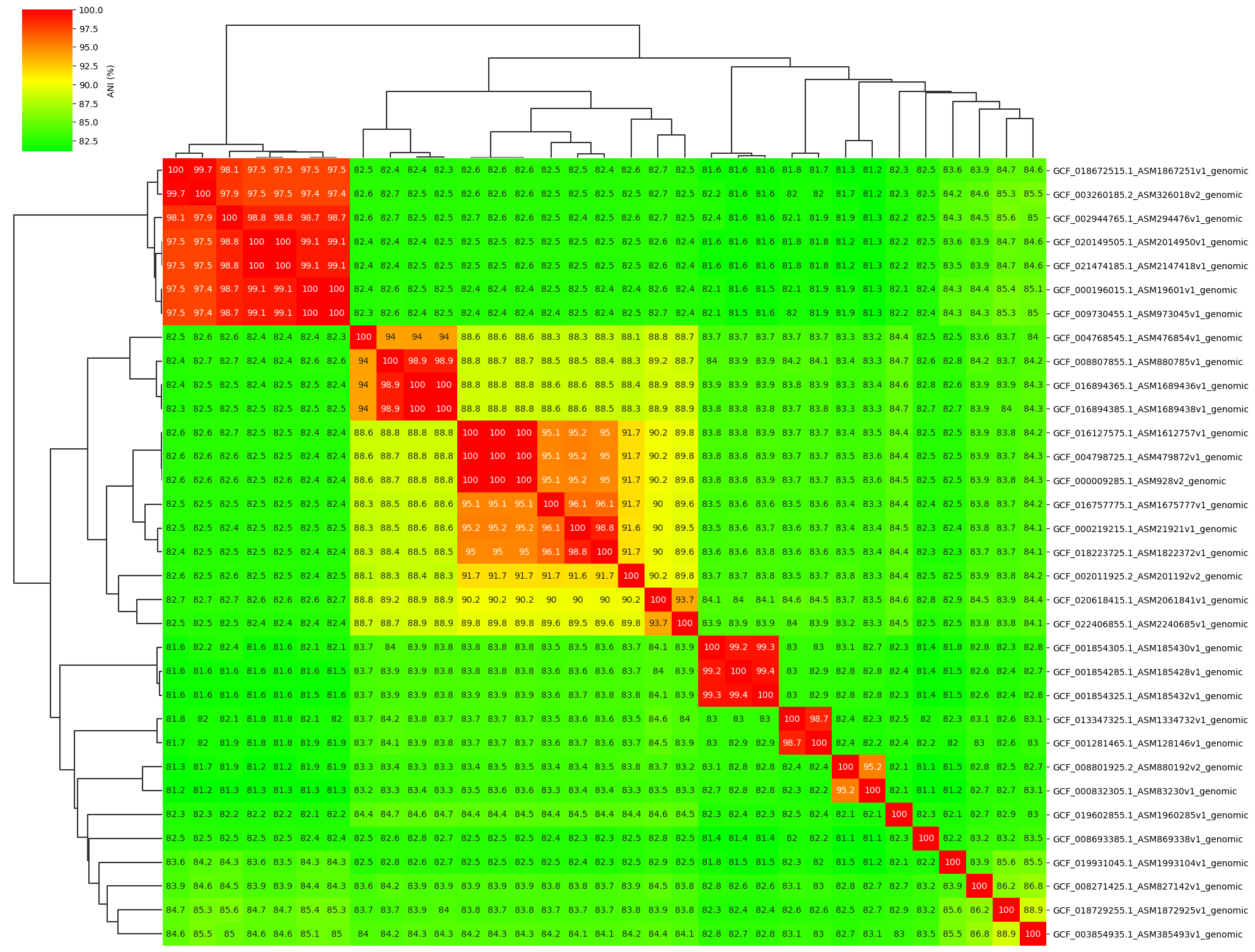
Fig1. ANI clustermap between all-vs-all 33 genomes.
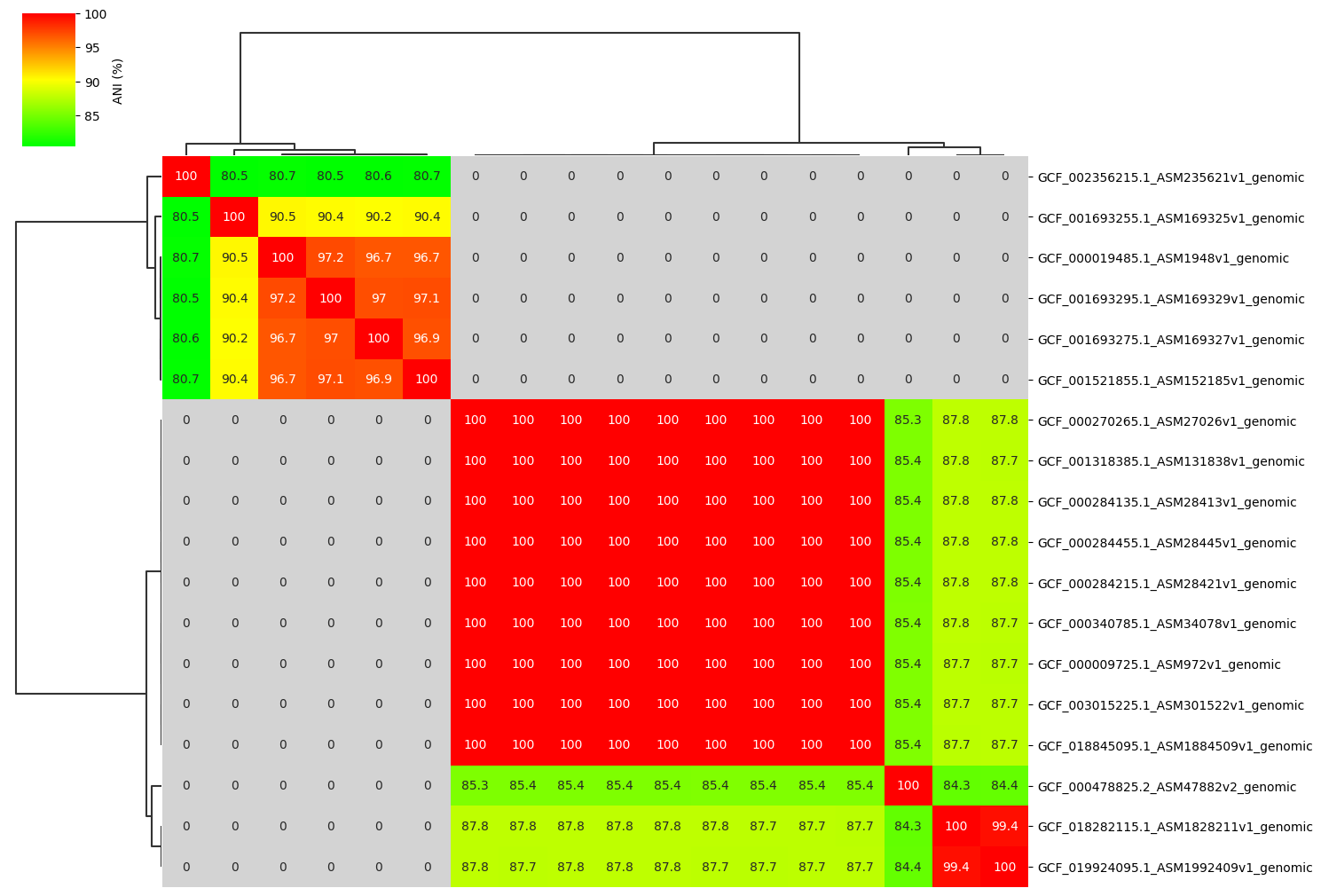
Fig2. ANI clustermap between all-vs-all 18 genomes. If no similarity detected by fastANI, filled in gray.
## Installation
ANIclustermap is implemented in Python3. [fastANI](https://github.com/ParBLiSS/FastANI) is required to calculate ANI.
**Install bioconda package:**
conda install -c bioconda -c conda-forge aniclustermap
**Install PyPI stable package:**
pip install aniclustermap
**Install latest development package:**
pip install git+https://github.com/moshi4/ANIclustermap.git
> :information_source: From ANIclustermap v1.2.0, ANI calculation with [skani](https://github.com/bluenote-1577/skani) is also supported.
> This is still an experimental implementation and is being tested with skani v0.1.0. Please install skani if needed.
## Workflow
Description of ANIclustermap's automated workflow.
1. Calculate ANI between all-vs-all microbial genomes by fastANI (or skani).
If no similarity detected by fastANI, NA is output. In that case, NA is replaced by 0.0.
If previous result available at the time of re-run, reuse previous result.
2. Clustering ANI matrix by scipy's UPGMA method.
3. Using clustered matrix, draw ANI clustermap by seaborn.
## Usage
### Basic Command
ANIclustermap -i [Genome fasta directory] -o [output directory]
### Options
-i I, --indir I Input genome fasta directory (*.fa|*.fna[.gz]|*.fasta)
-o O, --outdir O Output directory
-m , --mode ANI calculation mode ('fastani'[default]|'skani')
-t , --thread_num Thread number parameter (Default: MaxThread - 1)
--overwrite Overwrite previous ANI calculation result (Default: OFF)
--fig_width Figure width (Default: 10)
--fig_height Figure height (Default: 10)
--dendrogram_ratio Dendrogram ratio to figsize (Default: 0.15)
--cmap_colors cmap interpolation colors parameter (Default: 'lime,yellow,red')
--cmap_gamma cmap gamma parameter (Default: 1.0)
--cmap_ranges Range values (e.g. 80,90,95,100) for discrete cmap (Default: None)
--annotation Show ANI value annotation (Default: OFF)
-v, --version Print version information
-h, --help Show this help message and exit
### Example Command
7 genomes minimal dataset. Click [here](https://github.com/moshi4/ANIclustermap/wiki/dataset/minimal_dataset.zip) to download dataset (Size=3.6MB).
ANIclustermap -i ./minimal_dataset/ -o ./ANIclustermap_result
## Output Contents
ANIclustermap outputs 3 types of files.
- **`ANIclustermap.[png|svg]`** ([example1](https://github.com/moshi4/ANIclustermap/blob/main/example/output/05_normal_dataset/ANIclustermap.png), [example2](https://github.com/moshi4/ANIclustermap/blob/main/example/output/06_normal_dataset_annotation/ANIclustermap.png))
ANI clustermap result figure.
- **`ANIclustermap_matrix.tsv`** ([example](https://github.com/moshi4/ANIclustermap/blob/main/example/output/05_normal_dataset/ANIclustermap_matrix.tsv))
Clustered all-vs-all ANI matrix.
- **`ANIclustermap_dendrogram.nwk`** ([example](https://github.com/moshi4/ANIclustermap/blob/main/example/output/05_normal_dataset/ANIclustermap_dendrogram.nwk))
Newick format clustering dendrogram.
## Gallery
Example gallery of 33 genomes normal dataset.
If you want to try it for yourself, click [here](https://github.com/moshi4/ANIclustermap/wiki/dataset/normal_dataset.zip) to donwload dataset (Size=63.5MB).
**Normal parameter:**
ANIclustermap -i ./normal_dataset -o ./ANIclustermap_result \
--fig_width 15
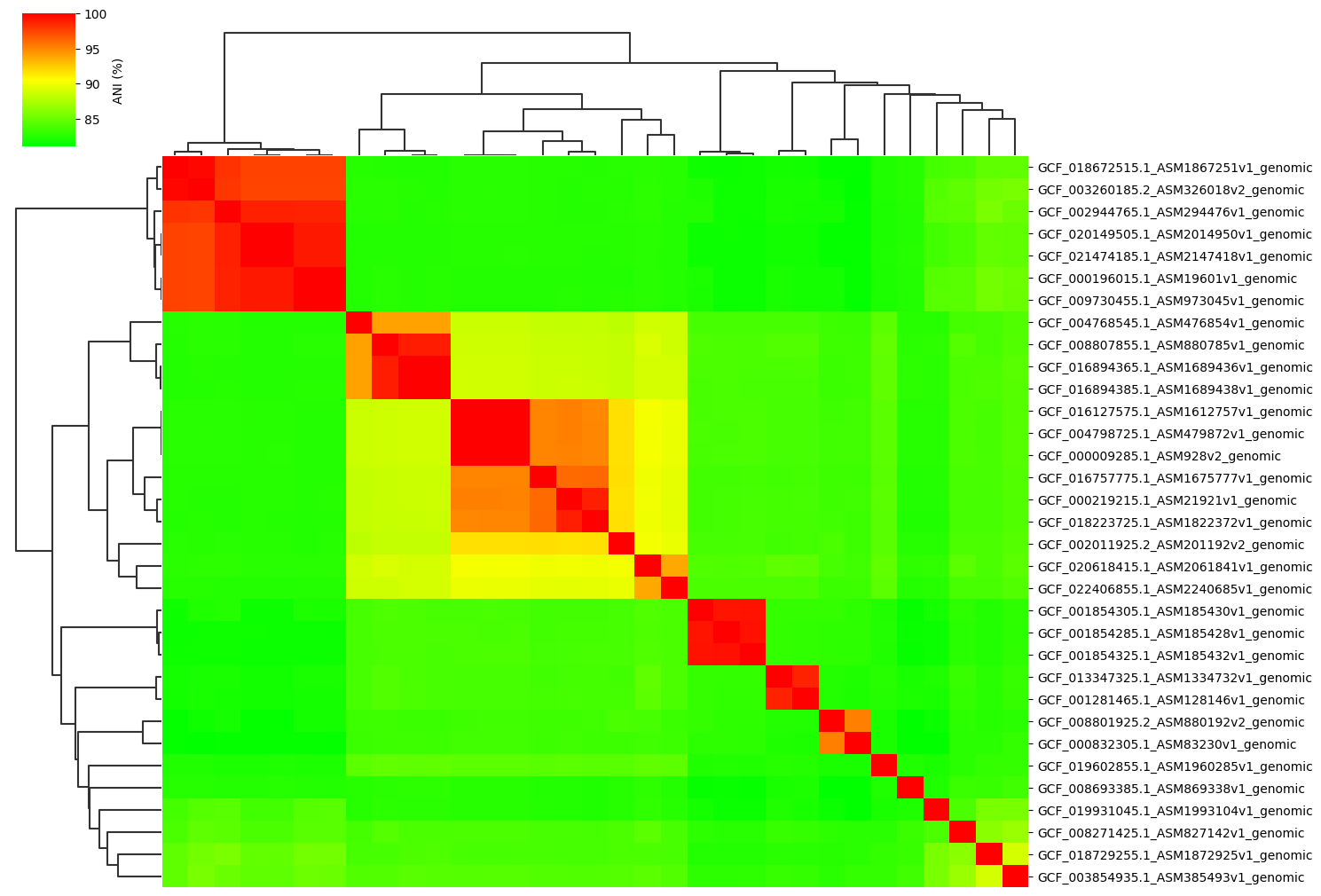
**Change cmap_gamma parameter:**
ANIclustermap -i ./normal_dataset -o ./ANIclustermap_result \
--fig_width 15 --cmap_gamma 0.5
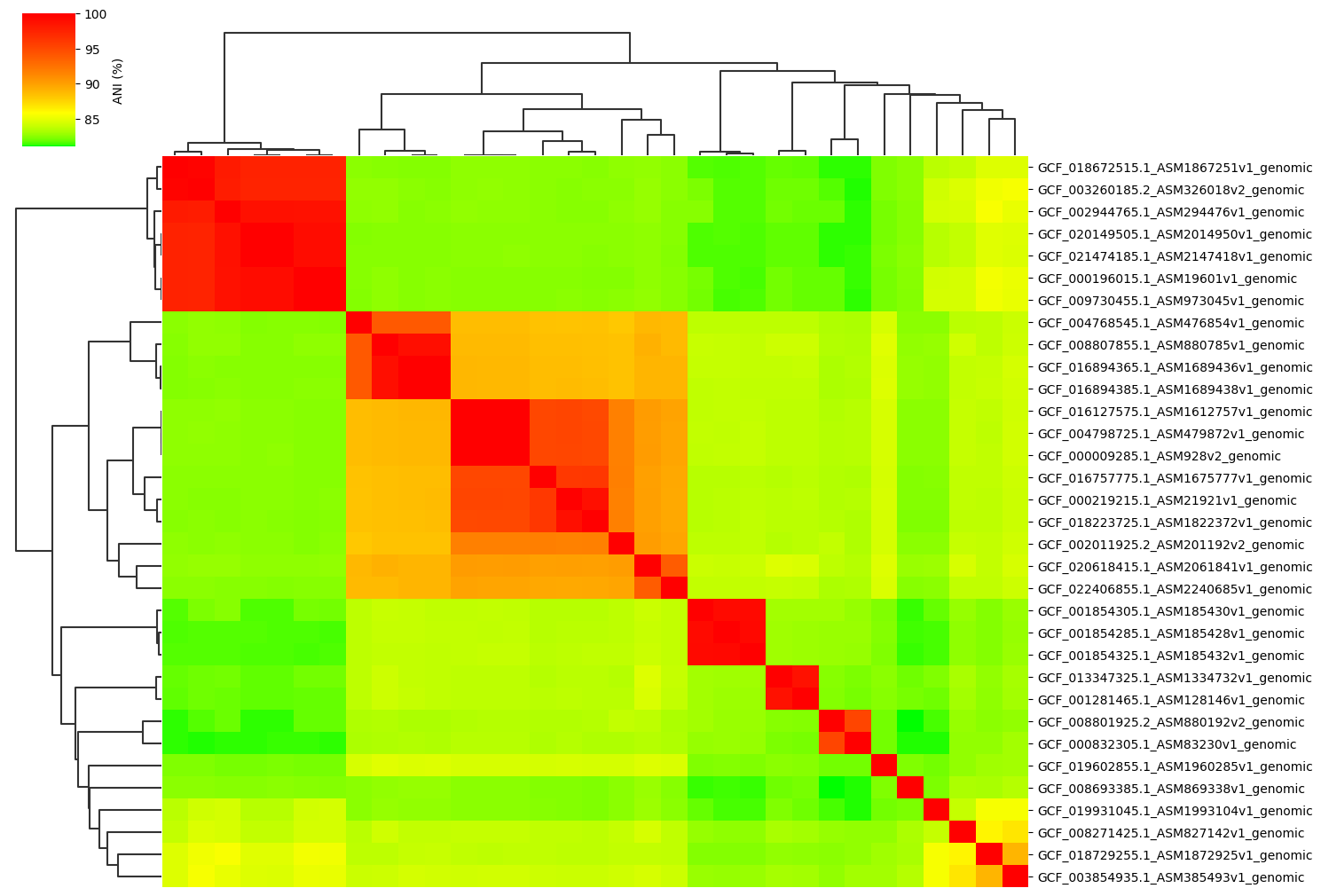
**Change cmap_colors(=white,orange,red) paramter:**
ANIclustermap -i ./normal_dataset -o ./ANIclustermap_result \
--fig_width 15 --cmap_colors white,orange,red
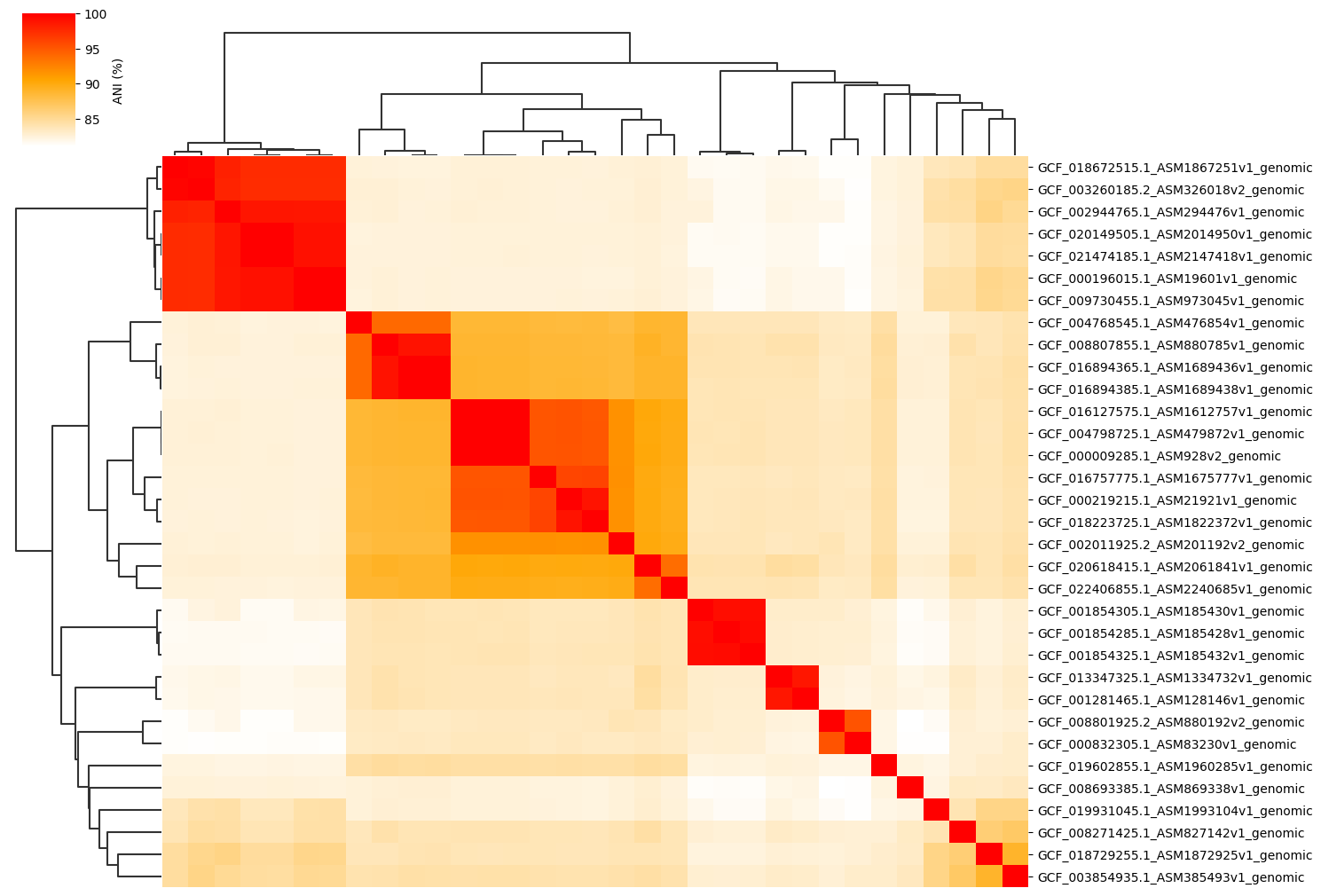
**Change cmap_ranges paramter:**
ANIclustermap -i ./normal_dataset -o ./ANIclustermap_result \
--fig_width 15 --cmap_ranges 80,85,90,92.5,95,97.5,100
> See [this issue](https://github.com/moshi4/ANIclustermap/issues/1) for more details.
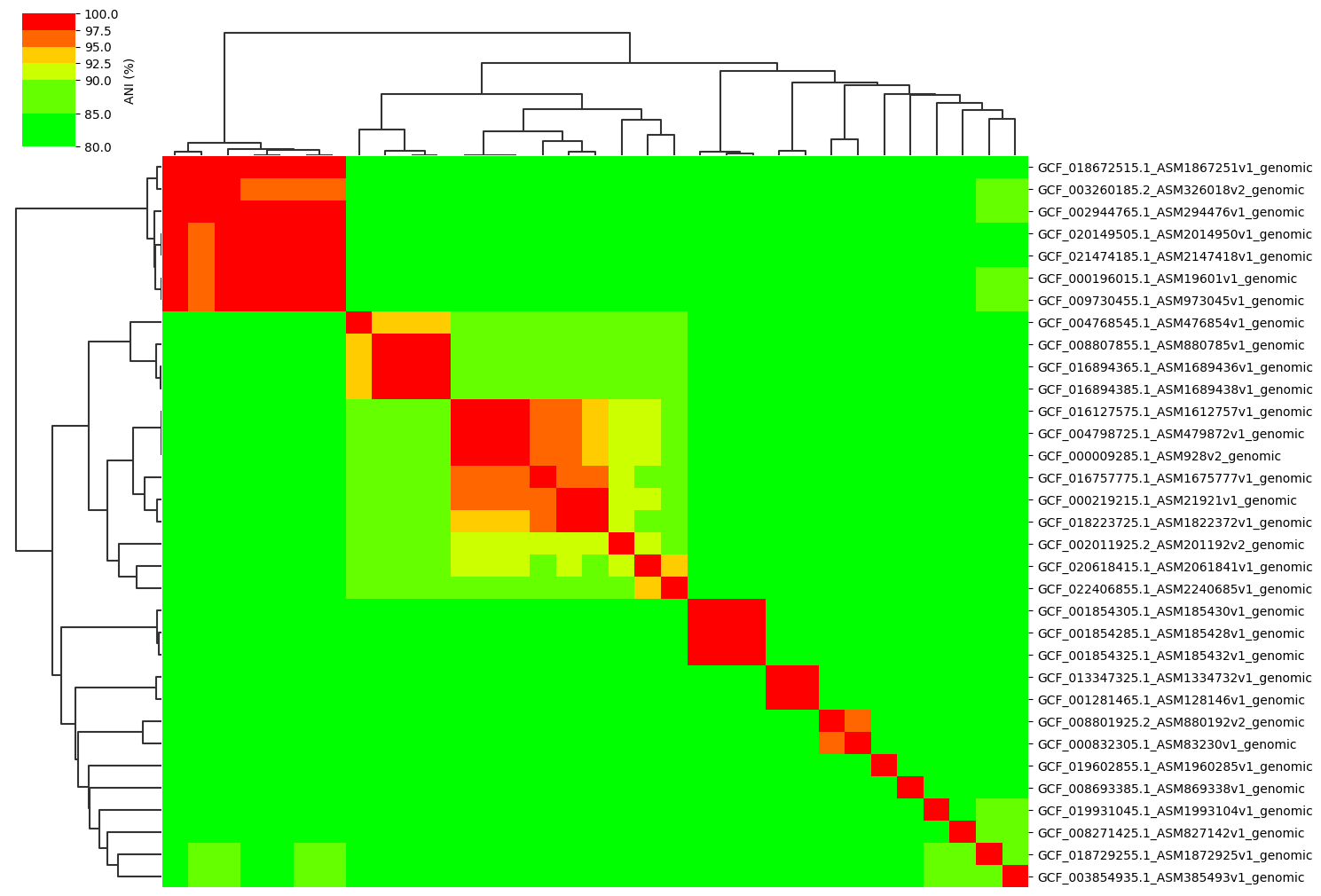
**Add ANI value annotation parameter:**
ANIclustermap -i ./normal_dataset -o ./ANIclustermap_result \
--fig_width 20 --fig_height 15 --annotation